In questo articolo descrivo come ho usato per molti anni il programma di visualizzazione molecolare Rasmol per delle esercitazioni pratiche di chimica generale presso l’Università dell’Aquila (Italia). Le esercitazioni consistevano nella visualizzazione di strutture cristallografiche di sistemi molecolari e nella misura di alcune proprietà geometriche. Per questo scopo è stato usato Rasmol controllato da un’interfaccia, scritta nel linguaggio Tcl/Tk, che permetteva di selezionare la struttura molecolare da visualizzare.
RasMol è uno tra i programmi più diffusi per la visualizzazione di strutture molecolari. Esso è liberamente distribuito e nel sito web (http://www.rasmol.org) è possibile ottenere il codice sorgente e gli eseguibili per i sistemi operativi Linux, MS Windows e Mac OS. Il programma ha un’ottima documentazione in lingua inglese. In questa pagina sono fornite le istruzioni necessarie per usare il programma per l’esercitazione. I lettori interessati sono comunque invitati ad esplorare le potenzialità di questo programma, installandolo sul proprio computer.
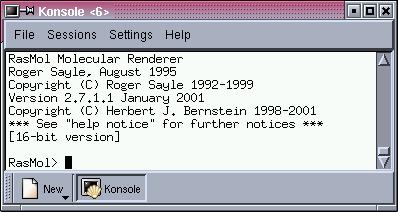
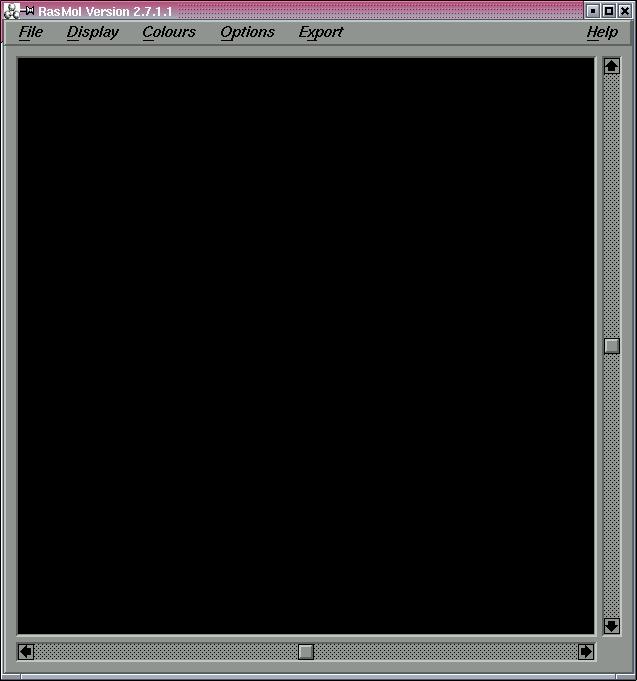
Per le esercitazioni, abbiamo usato dei PC provvisti di Linux OS. Il programma RasMol viene avviato dalla linea di comando di una finestra terminale digitando il comando rasmol. All’avvio, RasMol mostrerà una nuova finestra con sfondo nero e una barra di menu in alto, mentre nella finestra di lancio apparirà la linea di comando del programma con la scritta:
rasmol>
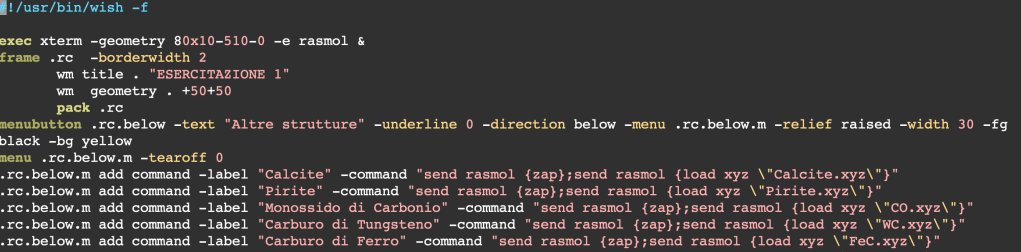
Nella figura sottostante sono rappresentate la finestra originaria di lancio del programma (A), da cui si impartiscono i comandi per RasMol, e la finestra di visualizzazione delle strutture (B). L’elenco giallo (C) è un programma di utilità, appositamente creato per il corso (e quindi non disponibile con il programma), per facilitare l’uso di RasMol durante le esercitazioni. Il programma fu scritto in Tcl/Tk e usava il comando send del Tcl per inviare, utilizzando la comunicazione interprocesso (IPC), comandi a RasMol. Quando RasMol si avvia su un sistema X Window, si registra con il X Window Server come un interprete Tcl. Da un’applicazione Tcl come ‘wish’, puoi usare il comando Tcl ‘winfo interps’ per determinare gli interpreti attualmente registrati su quel display. La prima istanza di RasMol si registra come ‘rasmol’, la seconda come ‘rasmol2’, la terza come ‘rasmol #3’ e così via. L’interprete Tcl può facilmente inviare un comando a RasMol utilizzando il comando integrato ‘send’. RasMol interpreta il parametro stringa del comando send non come una funzione Tcl da eseguire, ma come un comando RasMol. Quindi, digitando ‘send {rasmol} {background red}’ nell’interprete wish, farà cambiare colore alla finestra di visualizzazione di RasMol. RasMol eseguirà tutti i comandi in un ‘send’ prima di aggiornare lo schermo. Nella figura (D) che segue viene mostrata una parte del codice usato per l’interfaccia.

(D)
Le strutture da studiare potevano essere selezionate nella lista gialla, dove per ogni tipo di solido compare l’elenco di composti indicati nella Tabella I. Selezionato il composto, la sua struttura atomica comparirà nella finestra (B) di RasMol. La struttura può quindi essere ruotata o ingrandita muovendo il mouse e tenendo premuti i tasti del mouse e della tastiera secondo questa combinazione:
| Azione | Bottone del mouse e tasto della tastiera da premere |
|---|---|
| Ruota intorno a X, Y | Sinistro |
| Transla lungo X, Y | Destro |
| Ruota intorno a Z | Shift-Destro |
| Ingrandisci | Shift-Sinistro |
Alla voce Settings della barra del menu nella finestra (B) è possibile scegliere il tipo di azione che produce il doppio clic del tasto sinistro del mouse sulla struttura visualizzata. Nella tabella vengono descritte alcune di queste azioni.
| COMANDO | DESCRIZIONE |
| Pick off | Annulla l’azione di picking sulla struttura |
| Pick ident | Identifica la struttura cliccata dal mouse |
| Pick distance | Calcola distanza |
| Pick angle | Calcola angoli |
| Pick torsion | Calcola angoli di torsione |
| Pick label | Inserisce le etichette sugli atomi della struttura |
| Pick coord | Visualizza le coordinate atomiche dell’atomo |
| Pick bond | Seleziona un legame |
RasMol permette di creare o nuovi legami o rimuovere dei legami esistenti attraverso i seguenti comandi. Per creare un nuovo legame si usa il comando:
rasmol> bond atomo1 atomo2 +
dove atomo1 e atomo2 sono gli indici dei due atomi tra cui creare il nuovo legame che si ottengono cliccando con il mouse sugli atomi stessi. Mentre, per rimuovere un legame esistente si usa il comando:
rasmol> unbond atomo1 atomo2 .
VISUALIZZAZIONE E ANALISI DELLE STRUTTURE DI SOLIDI ATOMICI
Lo scopo dell’esperienza era quello di analizzare, usando RasMol, le proprietà geometriche e strutturali di composti appartenenti a diverse classi di solidi:
| TIPO DI SOLIDO | COMPOSTI |
| Ionico | 2 cristalli incogniti |
| Covalente | Diamante, grafite, silice |
| Metallico | Argento, Magnesio, Ferro |
| Molecolare | Ghiaccio, Iodio, Monossido di carbonio |
Per i vari composti veniva richiesto di analizzare alcune proprietà strutturali e geometriche. Per alcuni composti non veniva dato il nome e lo studente doveva eseguire i compiti elencati nell’esercitazione come descritto di seguito.
1. SOLIDI IONICI
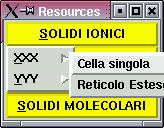
Selezionare con il mouse dal menu della finestra Resources: SOLIDI IONICI. Appariranno 3 composti incogniti XXX, YYY, ZZZ, per ognuno dei quali è possibile visualizzare la struttura della cella elementare o del reticolo esteso. Per ognuno dei tre composti, scegliere di visualizzare la cella elementare.
- Individuare il tipo di atomo che compongono ognuno dei tre composti,
- Per ognuno dei composti visualizzare il Reticolo Esteso
- Per ogni tipo di atomo presente nel composto calcolare:
- Contare gli atomi primi e secondi vicini
- Calcolare la distanza reticolare con gli atomi primi vicini
- Usare il valore della distanza reticolare e il numero di primi e secondi vicini per calcolare il valore approssimato della costante di Magdelung
2. SOLIDI COVALENTI
Selezionare con il mouse dal Menu della finestra Resources: SOLIDI COVALENT
Per i seguenti composti :
- Diamante
- Grafite
- Zolfo
- Quarzo
Osservate la cella elementare e il reticolo esteso.
Calcolare la distanza di legame per le seguenti coppie di atomi
- C – C (Diamante)
- C – C (grafite)
- S – S
- Si – O
Visualizzando il reticolo esteso della grafite calcolate la distanza tra i due piani selezionando due atomi di carbonio sovrapposti.
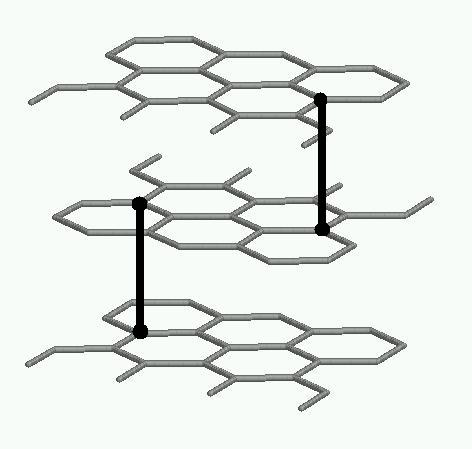
Misurare l’angolo di legame per le seguenti triplette di atomi
- C – C – C (diamante)
- C – C – C (grafite)
- S – S – S
- Si – O – Si
Per misurare gli angoli con RasMol:
- Selezionare dal Menu SETTINGS la scelta: Pick angle
- Fare un doppio click con il mouse sugli atomi che formano l’angolo di legame seguendo l’ordine della figura sottostante.
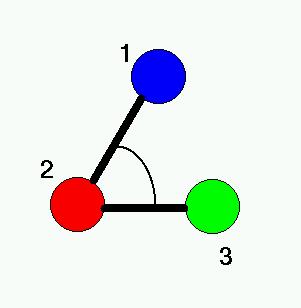
- Leggere il valore dell’angolo sulla finestra di visualizzazione o nella finestra di comando di RasMol.
2. SOLIDI METALLICI
Selezionare con il mouse dal Menu della finestra Resources: SOLIDI METALLICI
Per i seguenti metalli :
- Argento (Cella Cubica)
- Magnesio (Cella esagonale)
- Ferro (Cella Cubica a corpo centrato)
Osservate la cella elementare e il reticolo esteso.
- Contare gli atomi primi vicini per ogni metallo.
- Calcolare la distanza reticolare con gli atomi primi vicini.
2. SOLIDI MOLECOLARI
Selezionare con il mouse dal Menu della finestra Resources: SOLIDI MOLECOLARI
Calcolare le distanze di legame intra e intermolecolari
- Selezionare per il Ghiaccio, la scelta Cella elementare
- Per una coppia di due molecole di acqua calcolare le seguenti proprieta’ geometriche
- Distanze intramolecolari :
- O – H (intramolecolare)
- O — H (intermolecolare)
- O — O
- Angoli di legame:
- H – O – H (intramolecolare)
- O – H —- O (intermolecolare
